Ein schwieriges Unterfangen: die phylogenetische Analyse von SARS-CoV-2-Daten
-
Autor:
Isabel Lacurie
- Quelle:
- Datum: 13.08.2020
Computerwissenschaftler am HITS und KIT untersuchen die Probleme phylogenetischer Methoden bei der Analyse der Evolution des Coronavirus: viele Sequenzen, wenige Mutationen.
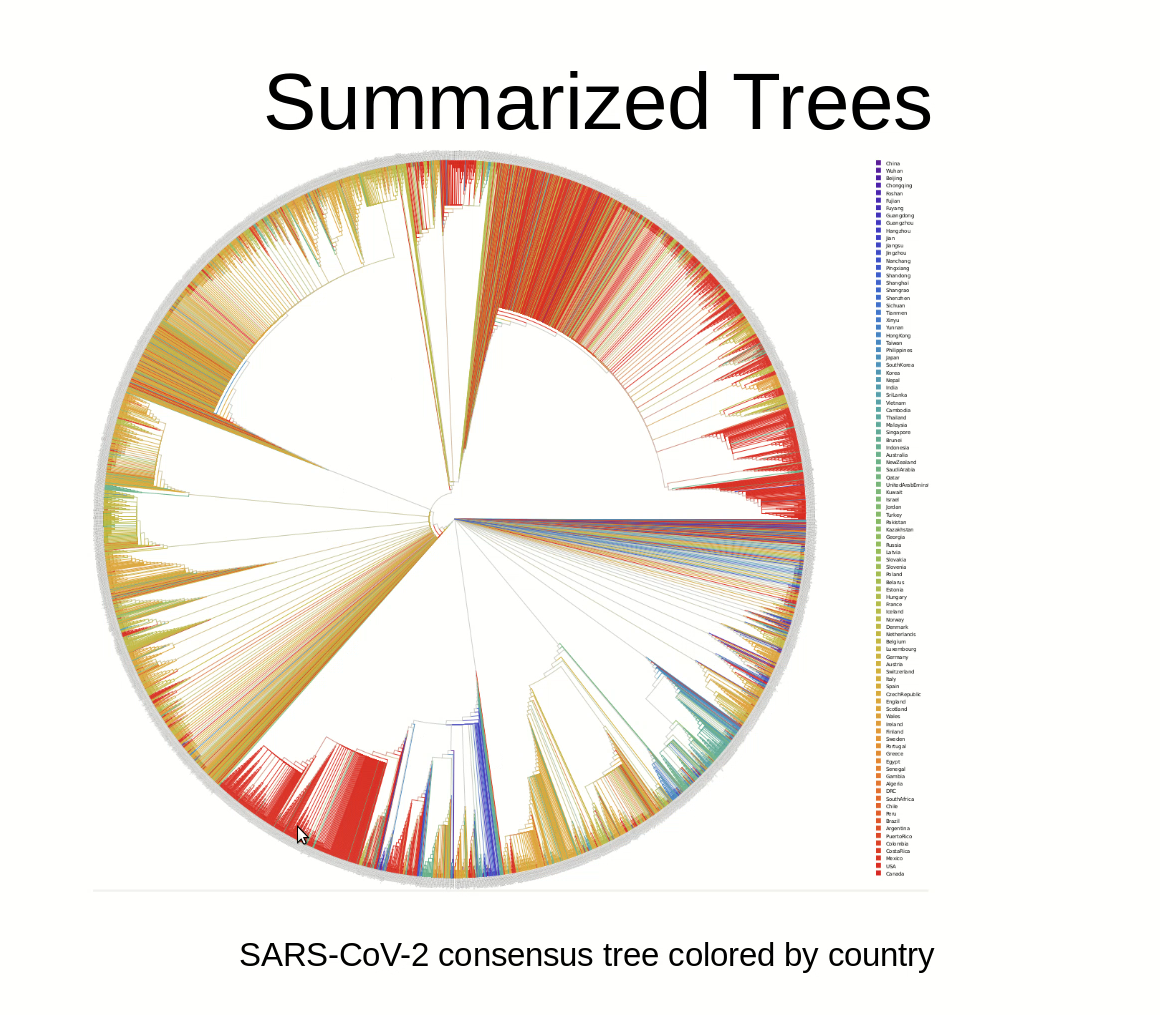
Tagtäglich werden zahlreiche wissenschaftliche Arbeiten zu verschiedenen Aspekten der Datenanalyse bei SARS-CoV-2 veröffentlicht, einschließlich eines laufend aktualisierten Stammbaumes. In einer als Preprint erhältlichen Studie analysierten Wissenschaftler/-innen der Forschungsgruppe Computational Molecular Evolution group (CME) am Heidelberger Institut für Theoretische Studien (HITS) und dem Institut für Theoretische Informatik am Karlsruher Institut für Technologie (KIT) nun alle Anfang Mai verfügbaren Virus-Sequenzen. Dabei stellten sie fest, dass es schwierig war, auf der Grundlage dieser Daten einen verlässlichen Stammbaum zu berechnen. Dies liegt vor allem an der großen Zahl von Sequenzen, die mit einer geringen Anzahl von Mutationen einhergeht. „Die Daten enthalten ein extrem schwaches Signal”, so CME-Gruppenleiter Alexandros Stamatakis.
Die Forscher/-innen fanden heraus, dass es nicht möglich war, die Wurzel des Stammbaumes verlässlich zu bestimmen – weder durch Hinzufügen von Fledermaus- und Schuppentier-Sequenzen zum Stammbaum noch durch die Anwendung neuester computergestützter Methoden auf den Stammbaum des Virus beim Menschen. Auch eine automatische Klassifikation der Sequenzen war nicht möglich, da die Sequenzen zu eng verwandt waren.
Die Wissenschaftler/-innen kommen zu dem Schluss, dass Stammbaumanalysen von SARS-CoV-2-Daten mit großer Vorsicht zu bewerten sind. „Man sollte keinesfalls Rückschlüsse aus nur einem einzelnen Baum ziehen”, so Alexandros Stamatakis.
Die Studie ist über den bioRxiv Preprint Server abrufbar:
Phylogenetic analysis of SARS-CoV-2 data is difficult. Benoit Morel, Pierre Barbera, Lucas Czech, Ben Bettisworth, Lukas Hübner, Sarah Lutteropp, Dora Serdari, Evangelia-Georgia Kostaki, Ioannis Mamais, Alexey M Kozlov, Pavlos Pavlidis, Dimitrios Paraskevis, Alexandros Stamatakis: https://www.biorxiv.org/content/10.1101/2020.08.05.239046v1
Professor Alexandros Stamatakis entwickelt Software zur Analyse großer biologischer Datenmengen. Sein Forschungsschwerpunkt ist die Entwicklung skalierbarer Methoden und Software zur Analyse molekularer Daten. Stamatakis´ Publikationen beeinflussten damit die Evolutionsbiologie der letzten Jahre nachhaltig. Im Zentrum stehen dabei die Entwicklung von Software zur Berechnung evolutionärer Stammbäume, die Evaluierung und der Einsatz neuer paralleler Rechnerarchitekturen, die Evolution von Krebszellen und die statistische Klassifikation von Darmbakterien. Alexandros Stamatakis entschlüsselte im Rahmen zweier internationaler Forschungsprojekte den Stammbaum der Vögel sowie den Stammbaum der Insekten. Er leitet seit 2010 die HITS-Forschungsgruppe „Computational Molecular Evolution“ und ist seit 2012 außerdem Professor für High Performance Computing in den Lebenswissenschaften am Karlsruher Institut für Technologie (KIT).